Das GHGA-Metadatenmodell unterstützt die wissenschaftliche Gemeinschaft dabei, die von ihr eingereichten Genomdaten umfassend zu beschreiben. Gleichzeitig hilft es Forschenden, relevante Daten zu finden und abzurufen. Um dies zu ermöglichen, konzentrieren wir uns darauf, die Metadaten auffindbar, zugänglich, interoperabel und wiederverwendbar (FAIR) zu machen. Dabei nutzen wir etablierte und weit verbreitete ontologische Konzepte und Terminologien, die wir in enger Zusammenarbeit mit unseren Partnern definiert haben.
Das GHGA-Metadatenmodell wird mit der Linked Data Modelling Language (LinkML) implementiert und ist im GHGA GitHub Repository frei zugänglich. Hier können Sie jede neue Version des Modells verfolgen sowie auf das Schema und automatisch generierte Excel-Vorlagen zugreifen.
Eine ausführliche Dokumentation des GHGA-Metadatenmodells und der dazugehörigen Hilfsmittel, einschließlich Beschreibungen des Modells selbst und der zugrunde liegenden Konzepte und Standards, finden Sie in der GHGA-Nutzer*innendokumentation.
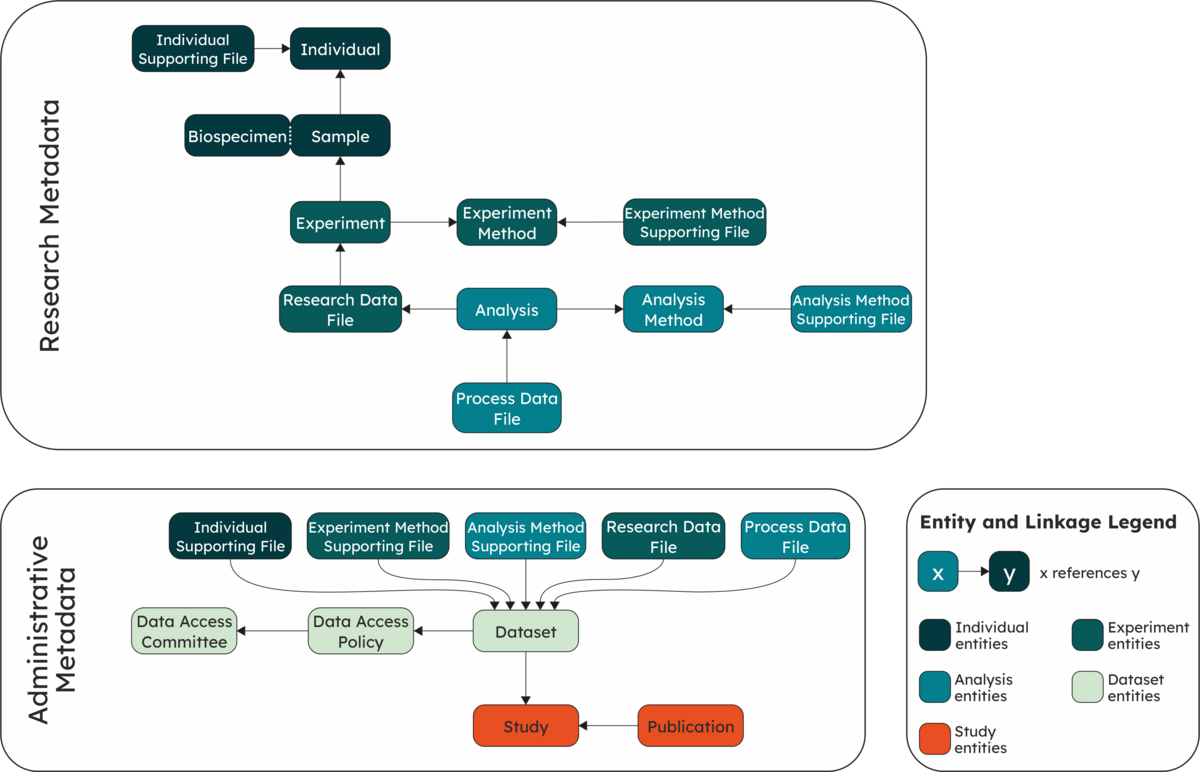
Die Modellklassen gewährleisten Interoperabilität und bieten gleichzeitig genügend Flexibilität, um verschiedene experimentelle Ansätze und Analysen abzudecken. Die Klassen sind nach Funktionalität gruppiert:
Die Forschungsmetadaten (Research Metadata) konzentrieren sich auf die Wiederverwendbarkeit und FAIRness der Daten, während die administrativen Metadaten die Bedingungen und das Komitee für den Datenzugriff beinhalten.
Forschungs-Metadaten (Research Metadata)
Administrative Metadaten
Die Metadaten können mithilfe des GHGA Submission Spreadsheet eingereicht werden. Dieses spiegelt das GHGA-Metadatenmodell wider und ermöglicht es dem GHGA-Datenportal, wertvolle Informationen über einen eingereichten Datensatz durch die Verknüpfung aller Kategorien anzuzeigen. Reichhaltig beschriebene Metadaten verbessern die Auffindbarkeit und erleichtern die Wiederverwendung der Daten innerhalb der Forschungsgemeinschaft.